同源建模
本课程旨在通过使用Swiss-Model在线工具对小鼠E-selectin蛋白进行同源建模,进而了解同源建模的基本概念和应用、了解Swiss-Model的功能和操作步骤。
1 获取模板蛋白序列
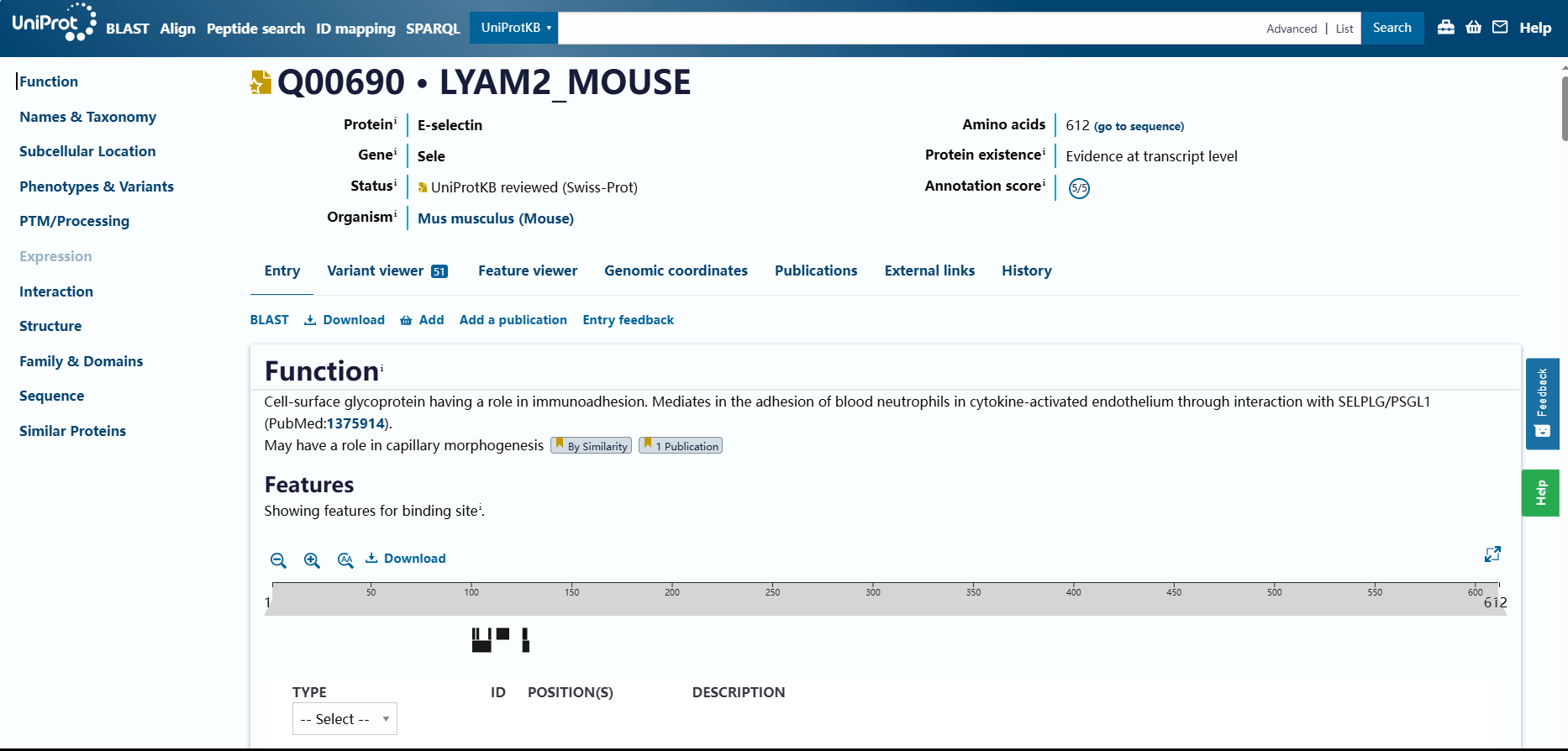 |
1. A. 在Uniport中搜索Mouse E-selectin即可获得相应信息,本课程直接提供了Q00690 · LYAM2_MOUSE的Uniport网页链接: Here.
|
 |
B、在该网页中直接点击“Download”
|
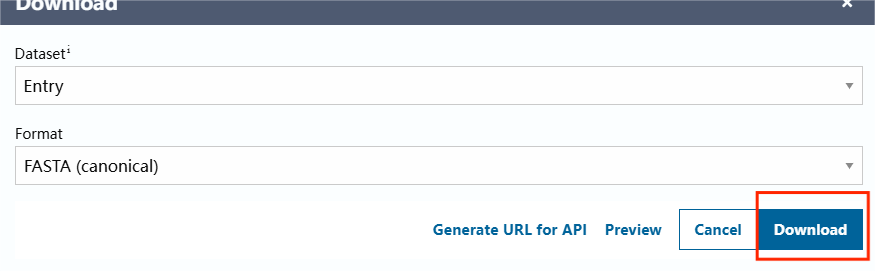 |
选择FASTA格式进行下载,选择好后直接点击Download进行下载” |
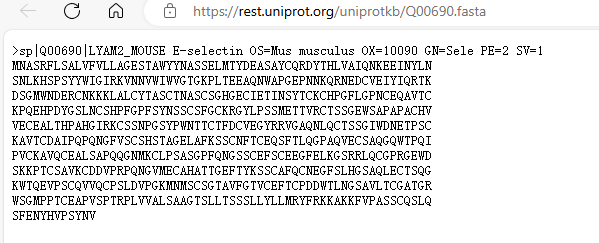 |
下载好的FASTA文件如图所示: |
这里直接给出了我们所需要的片段,大家可以在下一节时直接将其粘贴至对应窗口:
WYYNASSELMTYDEASAYCQRDYTHLVAIQNKEEINYLNSNLKHSPSYYWIGIRKVNNVWIWVGTGKPLTEEAQNWAPGEPNNKQRNEDCVEIYIQRTKDSGMWNDERCNKKKLALCYTASCTNASCSGHGECIETINSYTCKCHPGFLGPNCEQAVTCKPQEHPDYGSLNCSHPFGPFSYNSSCSFGCKRGYLPSSMETTVRCTSSGEWSAPAPACHVVECEALTHPAHGIRKCSSNPGSYPWNTTCTFDCVEGYRRVGAQNLQCTSSGIWDNETPSCKA
2 使用Swiss-Model进行同源建模
A. 搜索模板
 |
点击以下链接进入Swiss-Model Here |
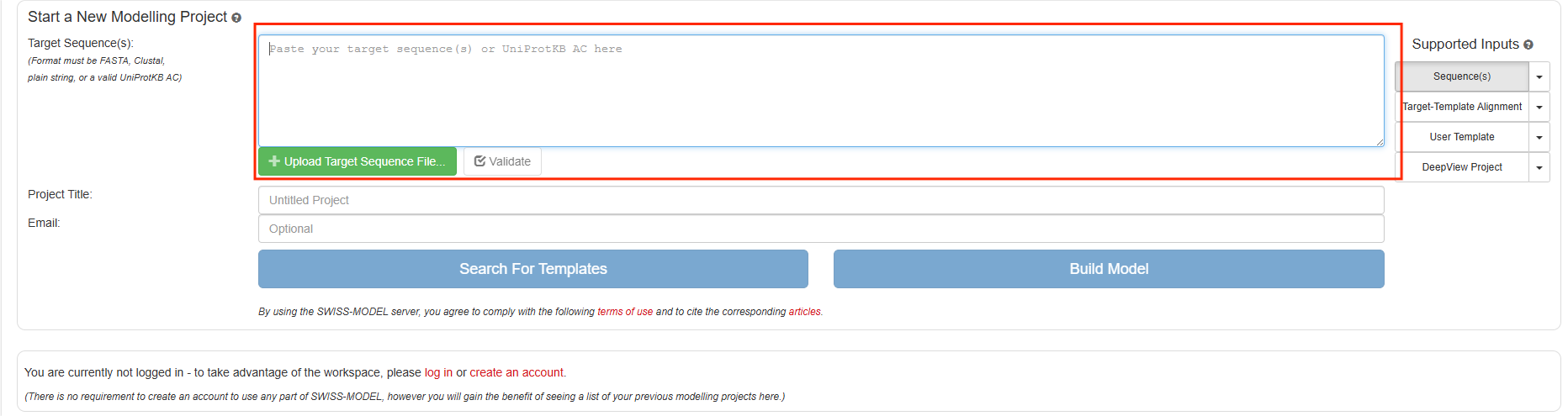 |
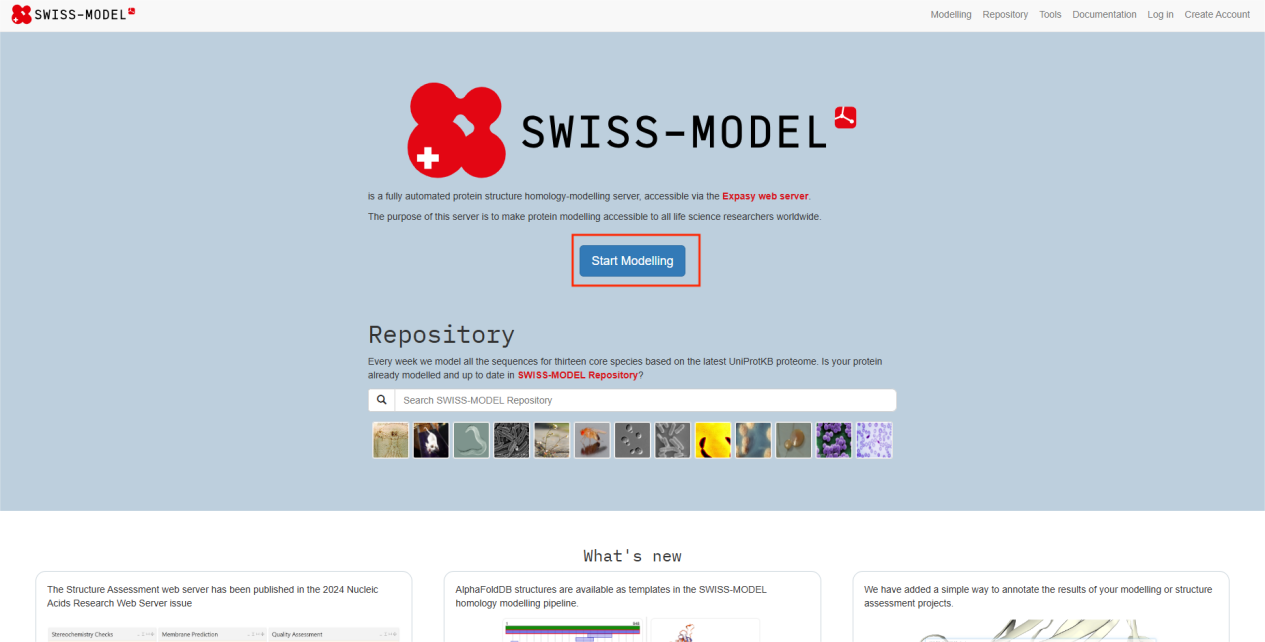
将上一步下载的目标蛋白Q00690 · LYAM2_MOUSE在教程中给出的的序列粘贴至“Target Sequence(s)”框中,这一部分是与human E-selectin crystal structure 对应的区间,所以仅取这一部分来进行模板的搜索 |
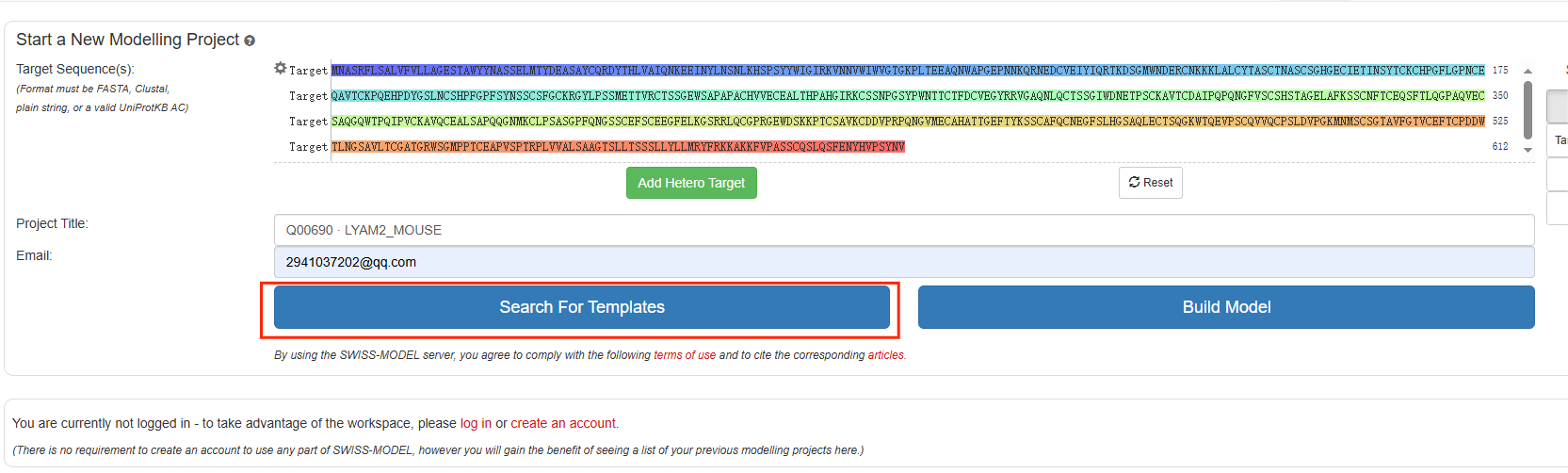 |
“Project Title”中填写项目名(任意,实例中为蛋白名称)、“Email”中填写自己常用的邮箱账号,点击“Search For Templates”按钮进行模板的搜索。 |
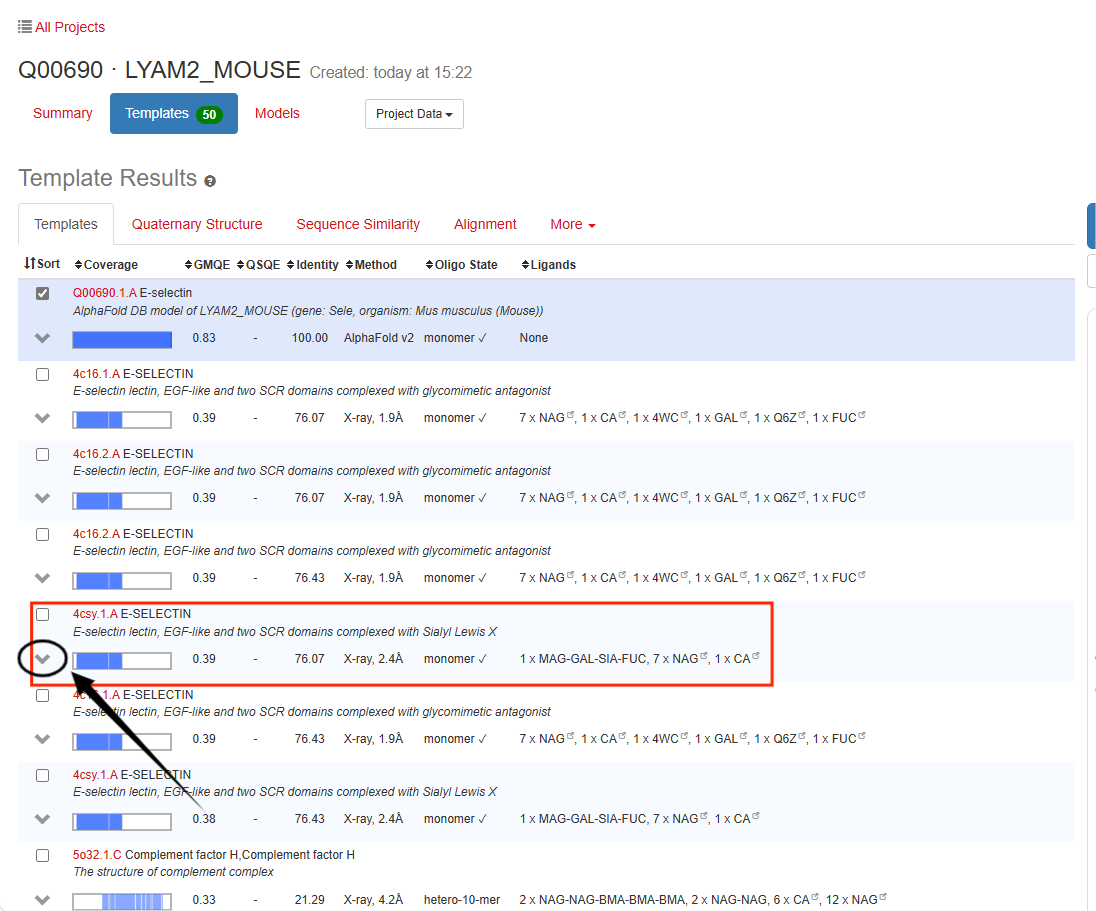 |
运行一段时间后,页面将给出模板结果列表,在其中选择⼀个蛋⽩做为模板,这⾥以4CSY为例,点击展开箭头将该条目展开。 |
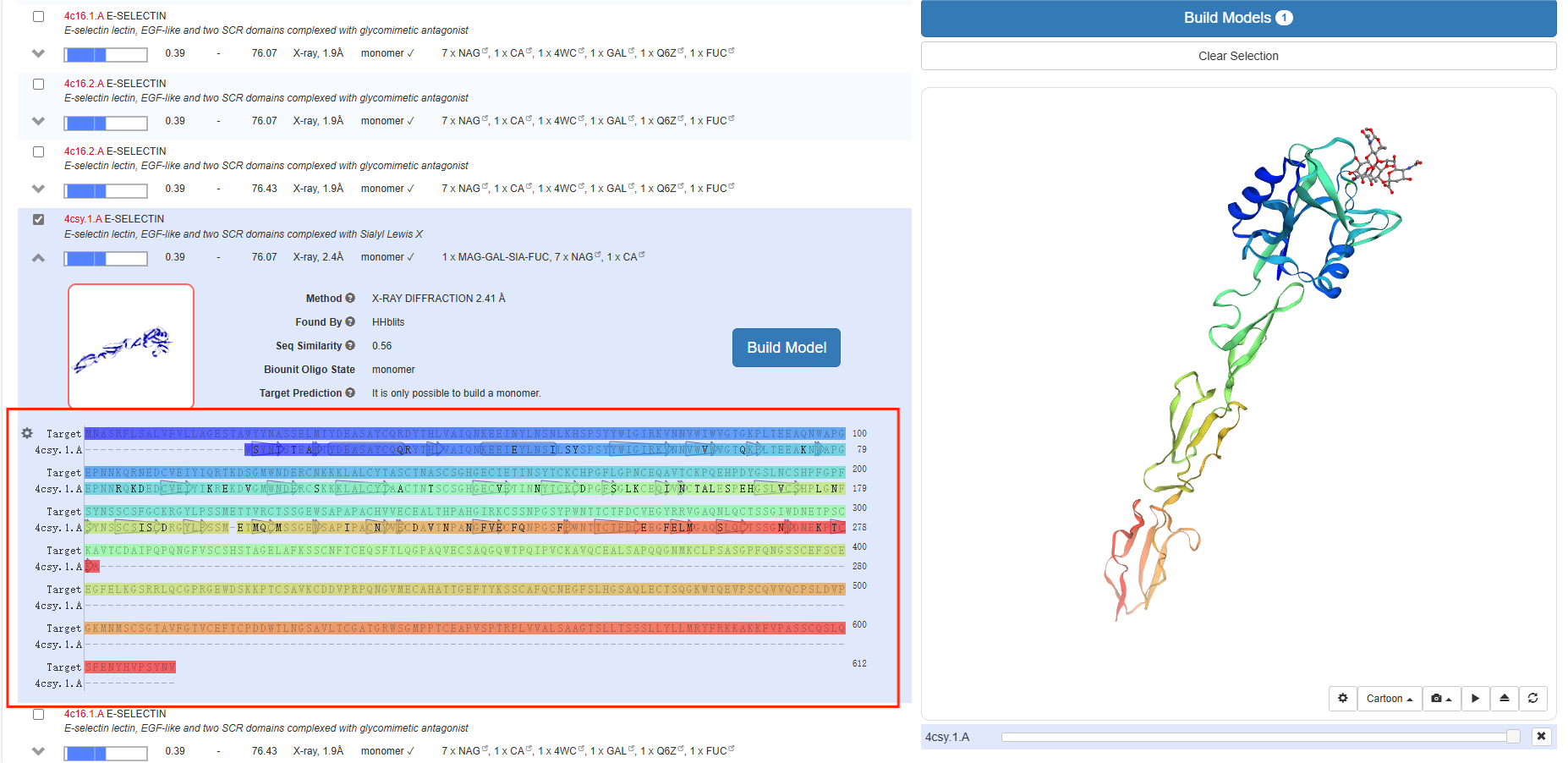 |
展开后,可以查看该模板蛋白与目标蛋白的序列比对信息,错配残基均已加黑标出。
界面右侧有其结构展示页面,可拖动观察结构细节。 |
 |
还可以通过齿轮标识进行进一步的比对分析 |
B. 同源建模
 |
点击“Build Model”按钮进行同源建模。 |
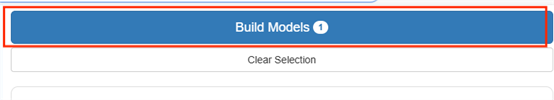 |
建模结束后,点击页面右上方蓝色按钮 |
 |
在跳转界面中展示了以4csy为模板进行同源建模结果的基本信息,点击“Stucture Assesment”按钮对该模型进行更进一步的评估 |
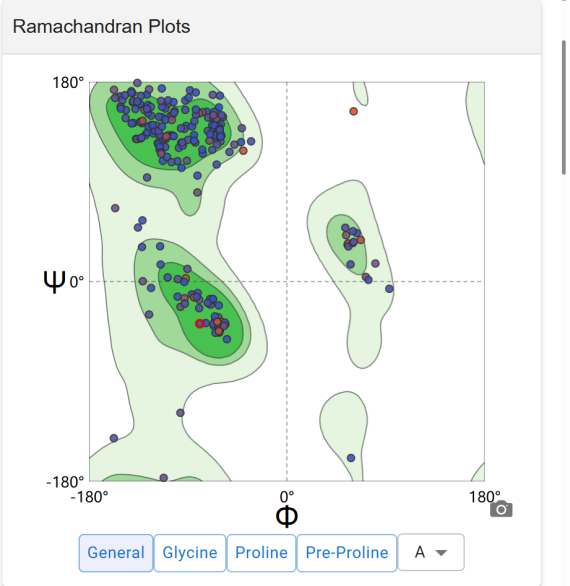 |
可获得该结构的拉氏图、MolProbity Results、QMEANDisCo以及比对结果等 |
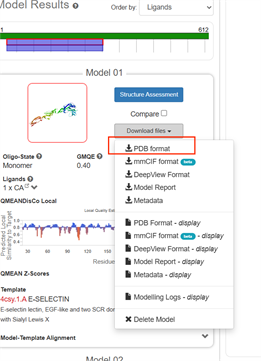 |
回到Model Results界面,点击“DownLoad files”按钮,选择“PDB Format”进行下载,并保存文件名为model.pdb. |
3. 截取模型的Lectin结构域
按照 VI编辑器->实例练习 中的方法截取模型的Lectin结构域与其钙离子。同样的,模型的前120个残基(residue ID 1-120)的构成了其lectin domain。 将model.pdb中的前120个残基(residue ID1-120)与钙离子粘贴至homo_model.pdb中,更改residue ID以及atom ID,同样的方法将钙离子更改保存为Mg+,并在蛋白与离子之间加上TER。
注:截取的residueid并不一定为1-120,例如,如果模型的residue number 从 22 开始,则截取的部分 residue number 为 22 -141;有时模型缺失头尾残基,建议导入VMD观察后决定截取范围
另外,使用以下命令将homo_model.pdb中的residue name “CYS”更改为“CYX”
注:“:1,$s/[space]CYS[space]/[space]CYX[space]/g
保存并退出vim。
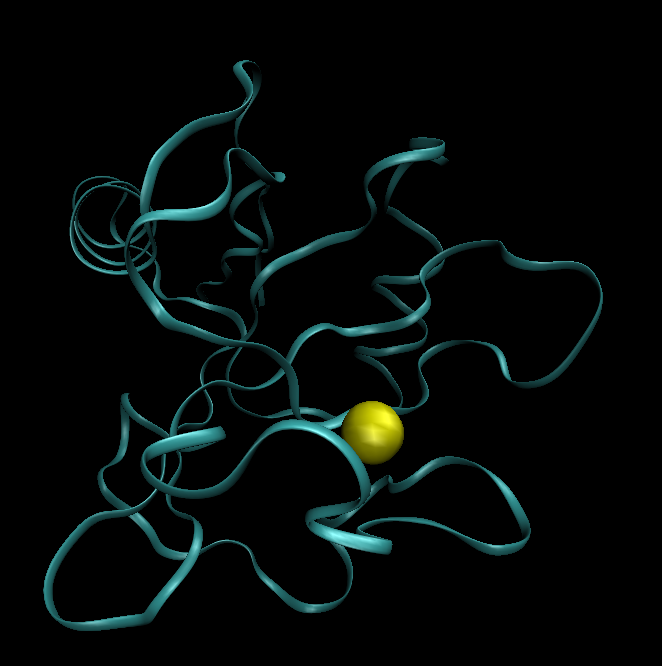 |
同样的方法将homo_model.pdb导入VMD观察其结构 |
Next

















