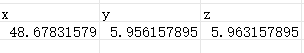分子对接
在上一章节中,我们使用4csy的human E-selectin 为模板,通过Swiss-Model构建出了mouse E-selectin。
在本章节中,我们将使用AutoDockTools研究mouse E-selectin与其配体sLex的结合过程,获得其复合物结构。
1 分子坐标叠合
首先,将 mouse E-selectin 与 human E-selectin - sLex 复合物中的蛋⽩进⾏叠合,可以获得 sLex 在mouse E-selectin 中的结合构象。
A.导入两个分子
 |
参考本课教程《VMD》部分,将crystal_human_E-selectin_sLex.pdb与homo_model.pdb导入至VMD。先点击VMDMain页面中File->New Molecular,导入crystal_human_E-selectin_sLex.pdb;导入成功后再次点击File->New Molecular,导入homo_model.pdb
导入成功之后VMD Main窗口显示如下: |
B. 进行分子叠合
 |
点击 Extensions->Analysis->RMSD Calculator |
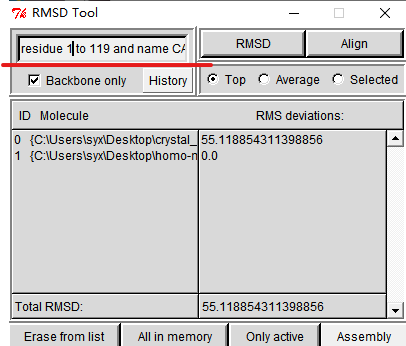 |
点击 选择参考模型为top(crystal_human_E-selectin_sLex.pdb),并选择你human E-selectin 对应的模型(crystal_human_E-selectin_sLex.pdb),残基选择设为residue 1 to 119 and name CA |
接下来,点击“RMSD”按钮,查看此时RMSD值
下面,点击“ALIGN”按钮,进行叠合,此时继续查看RMSD值。在该步骤中,若RMSD值变化很小也属正常情况
C. 在OpenGL Display中查看叠合效果
 |
在VMD Main->Graphics->Representations将crystal_human_E-seletin.pdb的参数设置为:
Coloring method -> “colorID”、Material -> AOChalky、Drawing Method->“NewCartoon”
同样的,将homo_model.pdb的参数设置为:
method -> “colorID”、Material -> AOChalky、Drawing Method->“NewCartoon”
两个模型设置为不同的颜色
|
D.导出叠合过后的mouse E-selectin的新pdb文件
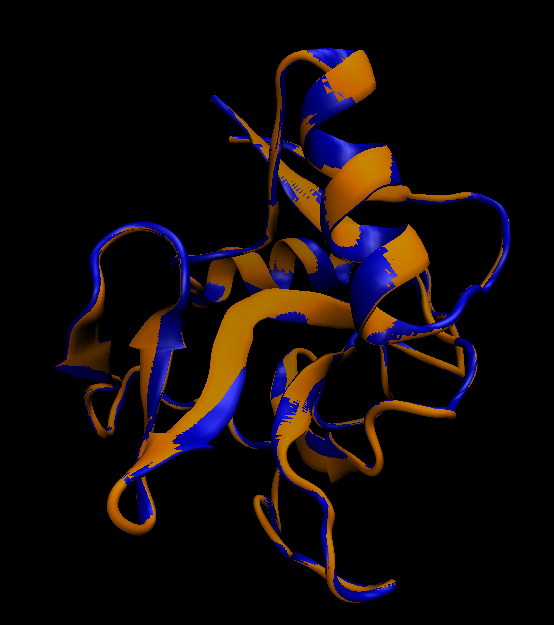 |
最后在OpenGL Display中查看叠合效果,可以看到,经过superimpose后,两个分子几乎已经在该坐标空间内叠合。 |
 |
在VMD Main中选择homo_model.pdb文件并鼠标右击,在弹出的窗口中选择Save Coordinates |
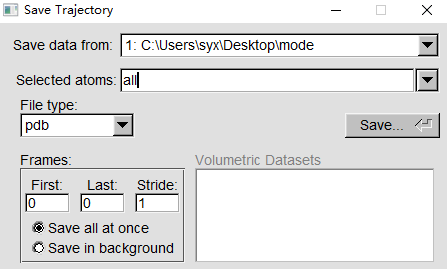 |
在弹出的窗口中,“Save data from”选择model_lectin.pdb文件、“Selected atoms”选择all、“File type”选择pdb,设置好之后按“Save”按钮,之后选择要保存的文件路径进行保存,示例中该文件名设置为superim_mouse_E-selectin.pdb。 |
E.合并鼠E-selectin与配体sLex获得其复合物
直接使用以下命令将superim_mouse_E-selectin.pdb的第一行至倒数第二行、crystal_human_E-selectin_sLex.pdb的配体内容重定向至新文件superim_mouse_E-selectin_sLex.pdb中,并在离子与配体之间加上“TER”。
$sed -n '1,$p' superim_mouse_E-selectin.pdb | tac | sed '1d' | tac > superim_mouse_E-selectin_sLex.pdb
$echo "TER" >> superim_mouse_E-selectin_sLex.pdb
$sed -n ‘1009,1070p’ crystal_human_E-selectin_sLex.pdb >> superim_mouse_E-selectin_sLex.pdb
2.下载AutoDockTools
A. 下载安装
这里直接提供了安装在不同平台上的mgltools安装包,请同学选择进行下载安装
Windows:windows
Mac:MAC
Linux:Linux
更多版本,请点击以下链接进行查看:mgltools
安装按照指引即可。
B. 准备蛋白
 |
在AutodockToos窗口中点击File -> Read Molecular,选择上一步准备好的superim_mouse_E-selectin.pdb文件进行导入 |
 |
导入完成后可视化界面显示如下 |
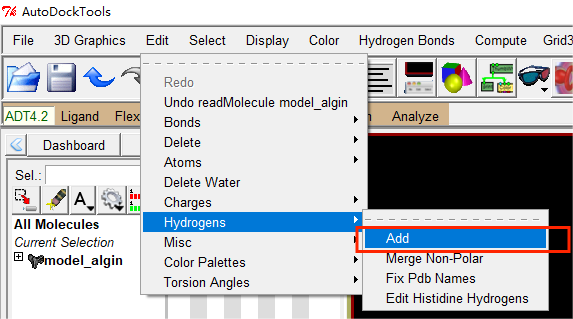 |
接着点击Edit->Hydrogens->add进行加氢 |
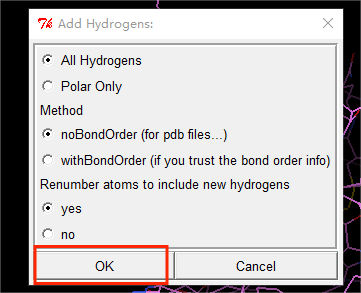 |
在弹出的窗口中点击“OK” |
 |
加氢完成后可视化界面显示如下 |
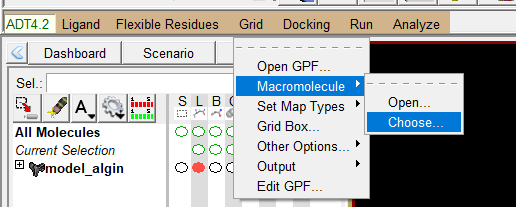 |
接下来,点击Grid->Macromolecule->choose…… |
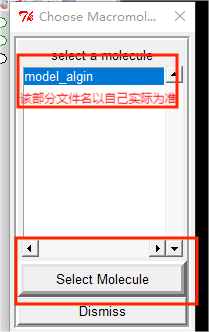 |
在弹出的窗口中选择蛋白模型,后点击“Select Molecule” |
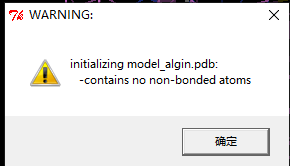 |
若弹出Warning警告框,点击确定即可 |
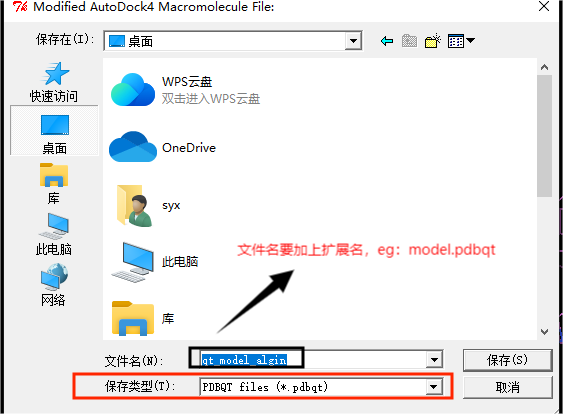 |
在弹窗中选择路径保,存为model.pdbqt文件 |
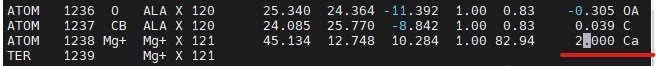 |
接下来,用vim打开model.pdbqt,找到其文件末尾的Mg+,将其电荷(倒数第二列所对应的数值)改为2.000 |
C.准备配体
 |
点击Ligand->Input->Open,在弹窗中选择sLex.pdb |
 |
在弹窗中将文件类型改为*.pdb,选择sLex.pdb |
 |
弹窗点击确定 |
 |
点击Ligand->Torsion Tree->Detect Root……进行软件自动判定配体root |
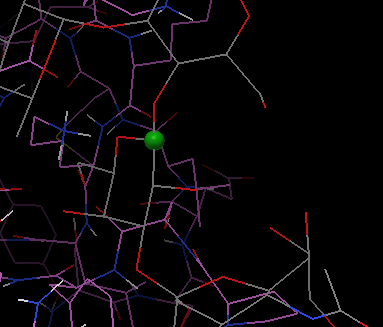 |
这时,出现的绿色小球即为分子Root |
 |
接下来,点击Ligand -> Torsion tree -> choose torsions查看配体分子的二面角 |
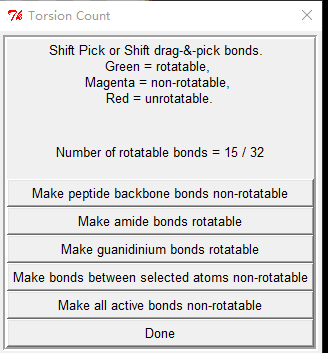 |
在弹窗中点击“DONE” |
 |
下面点击Ligand -> Torsion tree -> Set number of torsions 设置活性二面角数 |
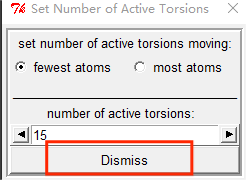 |
在弹窗中点击“Dismiss” |
 |
弹窗点击确定
最后,点击Ligand -> Output -> Save as PDBQT进行配体pdbqt格式文件的保存,文件名设置为ligand.pdbqt。 |
D.寻找分子对接中心
 |
将sLex.pdb导入excel中 |
 |
点击数据->分列 |
 |
选择固定宽度,在弹窗中连续点击下一步,最后点击完成。 |
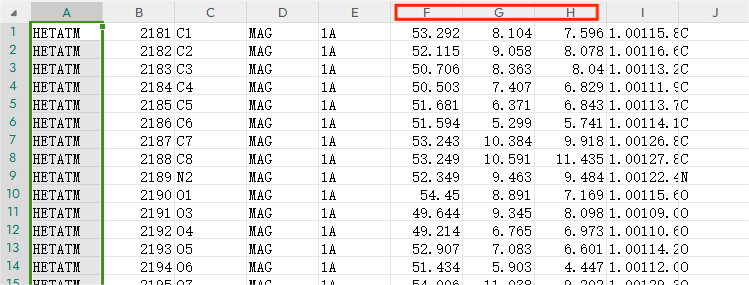 |
其中F、G、H列即为x,y,z坐标 |
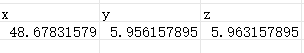 |
使用函数计算x,y,z的平均坐标,该做标即为分子对接的中心 |
E.进行分子对接
将上面两个文件移入autodock文件夹中
使用vi编辑器新建文件conf.txt,复制并粘贴以下内容到文件中,完成后保存.
receptor = model.pdbqt
ligand = ligand.pdbqt
out = out.pdbqt
log = out.txt
center_x = 48.678
center_y = 5.956
center_z = 5.963
size_x = 25
size_y = 25
size_z = 25
exhaustiveness = 80
num_modes =1
seed = 6
cpu = 1
现在,开始使用AutoDock Vina进行对接。out.pdbqt对应你保存的.pdbqt文件。新建文件run.com,复制并粘贴以下内容到文件中,完成后保存。
#!/bin/bash
/home/user/Downloads/autodock-vina/bin/vina --config conf.txt
使用以下命令给予文件执行权限,并运行:
$chmod +x run.com
$ ./run.com
对接结果在 out.pdbqt文件.
F. 对接结果的可视化
由于VMD无法识别.pdbqt格式文件,因此需要转为 *.pdb文件.
用vi编辑器打开文件,使用如下的命令得到可以被VMD识别的文件out.pdb.
并保证残基顺序为0SA,3LB,WYB,OME,0fA(与human E-selectin用相同的input),并在每个残疾之间加上TER。
将以下部分在粘贴至chuli2.sh
> docking_mouse_sLex.pdb
infilename="out.pdbqt"
residuename="0SA 3LB WYB OME 0fA"
for i in $residuename;
do
grep $i "$infilename" >> "docking_mouse_sLex.pdb";
echo "TER" >> "docking_mouse_sLex.pdb";
done
echo "END" >> "docking_mouse_sLex.pdb"
保存并退出vim,至终端输入以下命令运行chuli2.sh
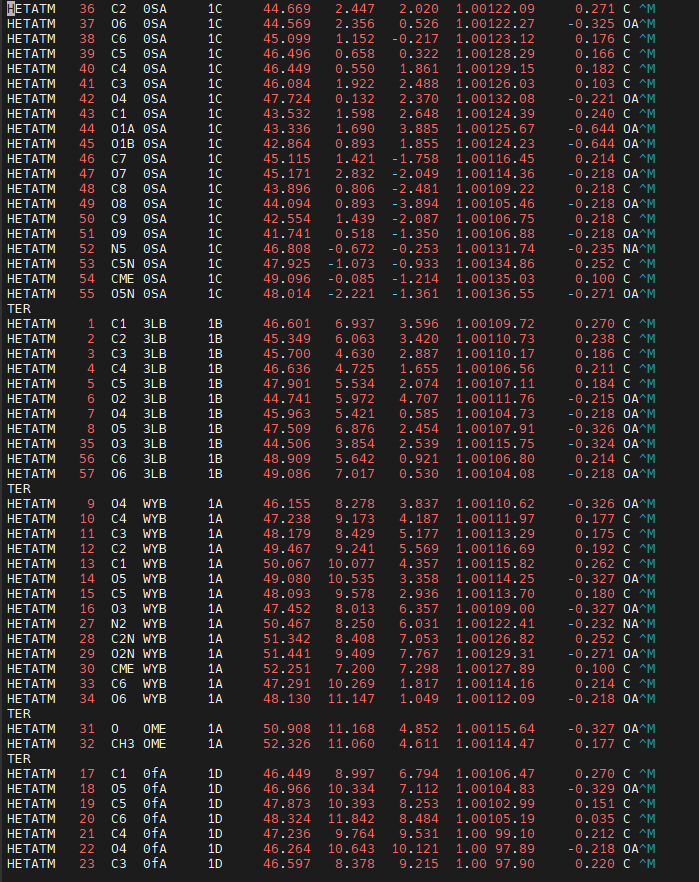 |
运行之后,当前文件夹下会出现docking_mouse_sLex.pdb文件,使用vim打开该文件. |
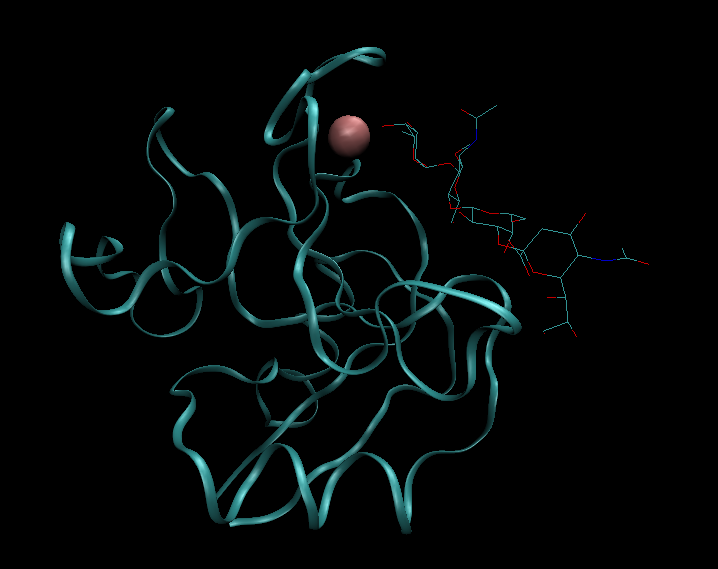 |
打开VMD, VMD Main->File->New Molecule首先加载刚刚保存的蛋白pdb文件(superim_mouse_E-selectin.pdb)。
然后将保存的配体结果文件(docking_mouse_sLex.pdb)作为New Molecule载入。可以看到该配体与蛋白结合的复合物。 |
VMD Main->Graphics->Representation,选择配体并通过改变Coloring Method和Drawing Method使其与蛋白区分开.
G.合并蛋白与doking所得的配体,获得复合物pdb文件
直接使用以下命令将superim_mouse_E-selectin.pdb的第一行至倒数第二行、docking_mouse_sLex.pdb的全部内容重定向至新文件docking_mouse_E-selectin_sLex.pdb并在离子与配体之间加上“TER”。
$sed -n ‘1,1008p’ superim_mouse_E-selectin_sLex.pdb > docking_mouse_E-selectin_sLex.pdb
$cat docking_mouse_sLex.pdb >> docking_mouse_E-selectin_sLex.pdb
接下来,去掉最后两列的元素与电荷(pdb文件中x、y、z坐标之后的列全部删去。推荐使用VISUAL BLOCK模式选中后直接按“d”键删除)
 |
处理好的文件部分展示如下:更改后的文件部分展示(图中展示了一些需要注意的点):
注意!如果受体蛋白的residue id 不是1-120,则离子与配体也应该做出相应改变,即按照蛋白->离子->配体的顺序直接顺序即可 |
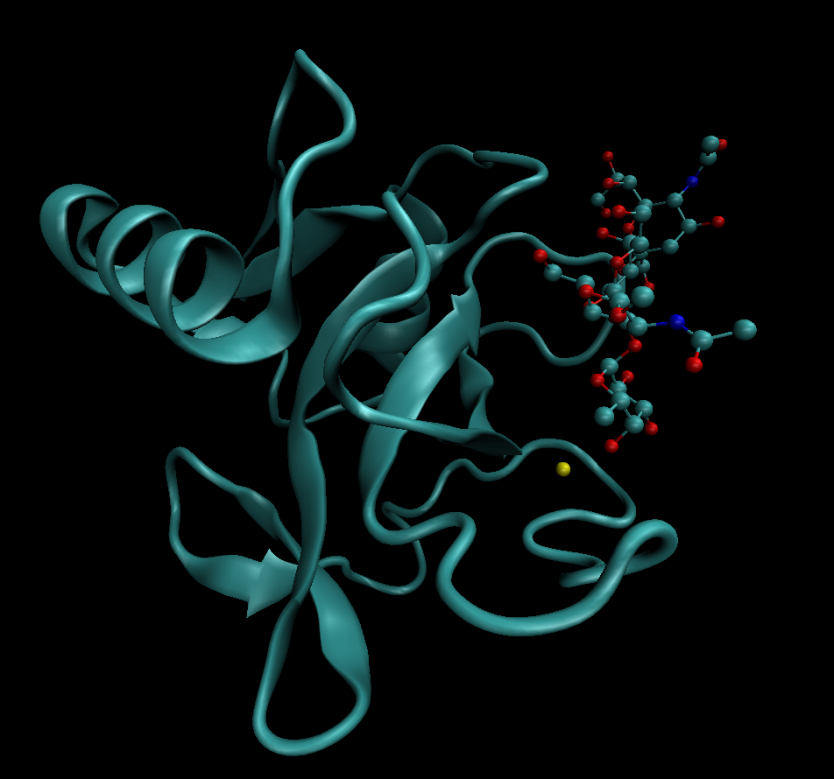 |
将docking_mouse_E-selectin_sLex.pdb导入VMD进行可视化展示: |
PRE
NEXT