分子动力学模拟
通过以上实验,我们已经获得了
1.人E-selectin的pdb文件crystal_human_E-selectin.pdb
2.同源建模所得并经过分子叠合的小鼠E-selectin的pdb文件mouse_E-selectin.pdb
3.配体的pdb文件sLex.pdb
4.Docking获得的配体pdb文件docking_mouse_sLex.pdb
本实验需要三人为一组,一组中三人分别获得以下结构:
1. human E-selectin – sLex from crystal structure
(之前从Swiss中下载的4csy.1.pdb中截取的部分,即crystal_human_E-selectin.pdb与sLex.pdb的合并文件,直接使用vim将其复制粘贴至新文件夹,将新文件夹命名为crystal_human_E-selectin_sLex.pdb);
2. mouse E-selectin – sLex, sLex 的binding pose通过superimpose产生
(superim_mouse_E-selectin.pdb(由分子坐标叠合得到)与sLex.pdb的合并文件,命名该文件为superim_mouse_E-selectin_sLex.pdb);
3. mouse E-selectin – sLex, sLex 的binding pose通过docking得到
(superim_mouse_E-selectin.pdb与docking_mouse_sLex.pdb的合并文件,命名该文件为docking_mouse_E-selectin_sLex.pdb)。
在本节实验中小组内的每个同学分别用自己所对应的结构根据以下分子模拟教程进行结构优化。
1. 新建工作文件夹
在主目录下新建文件夹md_simulations。
$ cd
$ mkdir md_simulations
将分子对接的结构文件粘贴至该文件夹
$cp ../autodock/crystal_human_E-selectin_sLex.pdb .
$cp ../autodock/crystal_human_E-selectin.pdb .
$cp ../autodock/sLex.pdb .
2.使用AMBER的tleap程序构建TOP和CRD文件
用vim编辑器在 md_simulations 目录下新建文件tleap.in,将以下内容粘贴至其中,其中着重标出部分为需修改或核对的部分,本tleap文件内容以human E-selectin – sLex from crystal structure体系的复合物为例,导入的文件为crystal_human_E-selectin_sLex.pdb;其中bond关键字对应的数字部分应视每个导入的pdb结构不同而进行改变
source leaprc.GLYCAM_06j-1
source oldff/leaprc.ff99SB
loadamberparams frcmod.ions234lm_126_tip3p
mol = loadpdb ./crystal_human_E-selectin_sLex.pdb
# protein #
# di-sulfide bond #
#以下以bond关键字开头的语句代表整个大分子之间的连接部分
#这里标绿的部分是atom name为SG(硫原子)的residue ID,这里代表二硫键
bond mol.19.SG mol.117.SG
bond mol.90.SG mol.109.SG
#以下代表着配体四糖的糖苷键信息,为糖连接原子的residue ID。
bond mol.122.C2 mol.123.O3 # Neu5Ac - Core-2 Gal
bond mol.123.C1 mol.124.O4 # Core-2 Gal - GlcNAc
bond mol.126.C1 mol.124.O3 # Fuc - GlcNAc
bond mol.124.C1 mol.125.O # GlcNAc - OME
charge mol
addIons mol Na+ 0
addIons mol Cl- 0
#输出文件也应更改为对应体系的文件名。
saveamberparm mol complex_human_E-selectin_sLex.prmtop complex_human_E-selectin_sLex.rst7
savepdb mol complex_human_E-selectin_sLex.pdb
solvateOct mol TIP3PBOX 8.0
saveamberparm mol OCT_complex_human_E-selectin_sLex_WAT.prmtop OCT_complex_human_E-selectin_sLex_WAT.rst7
savepdb mol OCT_complex_human_E-selectin_sLex_WAT.pdb
quit
注!当该文件用于同源建模(superim_mouse_E-selectin_sLex.pdb)与分子对接(docking_mouse_E-selectin_sLex.pdb)的模型构建时,若前面构建的蛋白结构域的residue id 不是从1-120,则二硫键的部分需要进行对应的修改,建议通过vim命令行模式寻找SG的residue id(/ SG )
保存并退出后在终端运行以下命令
$ tleap -f tleap.in
输出的OCT_complex_human_E-selectin_sLex_WAT.pdb文件的顶部包含蛋白信息,后面是离子、配体和溶剂信息。每个部分由TER标识进行分隔。文件中第五列给出的是残基数。使用vi打开PDB文件并输入/WAT后会跳转到水分子首次出现的地方,可以看到残基数结尾和水分子开始数,输入:$跳转到文件结尾,可以看到水分子结尾数,记录下各部分残基数量范围. (Example) 非水残基范围(蛋白+离子+配体): 1-126, Water: 127-4021 注意,有时残基数会超过10000,到了10000会重新从0开始计数,具体可在pdb中查看。
注!当该文件用于同源建模(superim_mouse_E-selectin_sLex.pdb)与分子对接(docking_mouse_E-selectin_sLex.pdb)的模型构建时,若前面构建的蛋白结构域的residue id 不是从1-120,则蛋白、离子、配体、Water的残基范围都会有所改变,具体查看模型pdb
3.准备动力学输入文件
AMBER所需要的第三个文件是参数文件(*.in)。这个文件包含的是你模拟过程的一些参数,包括体系压力、温度、非成键相互作用距离以及模拟过程中那些原子需要固定等等,通过查看AMBER手册去熟悉这些参数。
使用vim在 md_simulations目录下新建以下文件,红色字体为文件名:
1.min.in
 |
&cntrl imin = 1, maxcyc = 10000, ncyc = 5000, cut = 8.0, ntxo = 2, ntwr = 500, ntpr = 500, ntr = 1, restraintmask = '!:WAT= & !@H=', restraint_wt = 5.0, &end |
2.relax.in
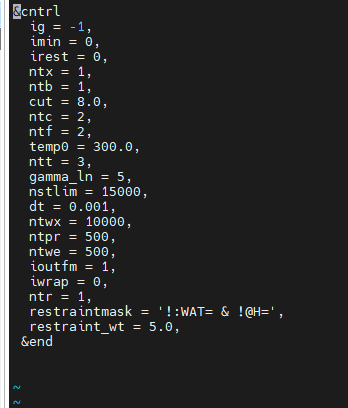 |
&cntrl ig = -1, imin = 0, irest = 0, ntx = 1, ntb = 1, cut = 8.0, ntc = 2, ntf = 2, temp0 = 300.0, ntt = 3, gamma_ln = 5, nstlim = 15000, dt = 0.001, ntwx = 10000, ntpr = 500, ntwe = 500, ioutfm = 1, iwrap = 0, ntr = 1, restraintmask = '!:WAT= & !@H=', restraint_wt = 5.0, &end |
3.min.in
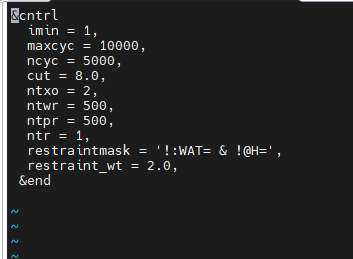 |
&cntrl imin = 1, maxcyc = 10000, ncyc = 5000, cut = 8.0, ntxo = 2, ntwr = 500, ntpr = 500, ntr = 1, restraintmask = '!:WAT= & !@H=', restraint_wt = 2.0, &end |
4.min.in
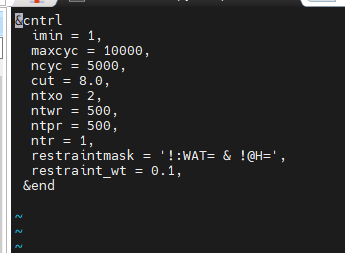 |
&cntrl imin = 1, maxcyc = 10000, ncyc = 5000, cut = 8.0, ntxo = 2, ntwr = 500, ntpr = 500, ntr = 1, restraintmask = '!:WAT= & !@H=', restraint_wt = 0.1, &end |
5.min.in
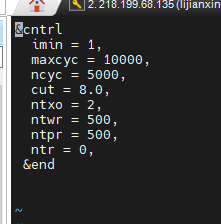 |
&cntrl imin = 1, maxcyc = 10000, ncyc = 5000, cut = 8.0, ntxo = 2, ntwr = 500, ntpr = 500, ntr = 0, &end |
6.relax.in
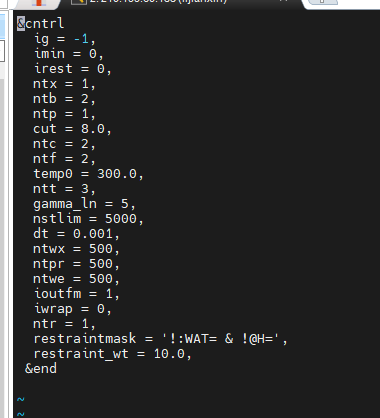 |
&cntrl ig = -1, imin = 0, irest = 0, ntx = 1, ntb = 2, ntp = 1, cut = 8.0, ntc = 2, ntf = 2, temp0 = 300.0, ntt = 3, gamma_ln = 5, nstlim = 5000, dt = 0.001, ntwx = 500, ntpr = 500, ntwe = 500, ioutfm = 1, iwrap = 0, ntr = 1, restraintmask = '!:WAT= & !@H=', restraint_wt = 10.0, &end |
7.relax.in
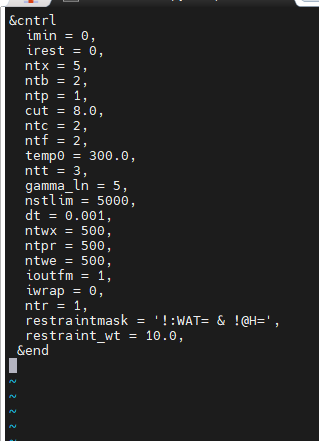 |
&cntrl imin = 0, irest = 0, ntx = 5, ntb = 2, ntp = 1, cut = 8.0, ntc = 2, ntf = 2, temp0 = 300.0, ntt = 3, gamma_ln = 5, nstlim = 5000, dt = 0.001, ntwx = 500, ntpr = 500, ntwe = 500, ioutfm = 1, iwrap = 0, ntr = 1, restraintmask = '!:WAT= & !@H=', restraint_wt = 10.0, &end |
8.relax.in
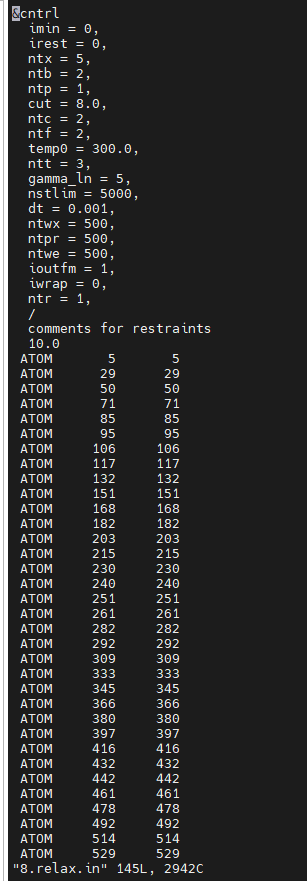 |
&cntrl imin = 0, irest = 0, ntx = 5, ntb = 2, ntp = 1, cut = 8.0, ntc = 2, ntf = 2, temp0 = 300.0, ntt = 3, gamma_ln = 5, nstlim = 5000, dt = 0.001, ntwx = 500, ntpr = 500, ntwe = 500, ioutfm = 1, iwrap = 0, ntr = 1, / comments for restraints 10.0 #该部分为所有“CA” 原子的atom number,此处仅为两个原子示例 ATOM 5 5 ATOM 120 120 END END |
(每一标亮部分都有8个letter,通过命令:grep CA OCT_complex_human_E-selectin_sLex_WAT.pdb > restraint.txt获取,再通过vim编辑器编辑成要求格式)
9.relax.in
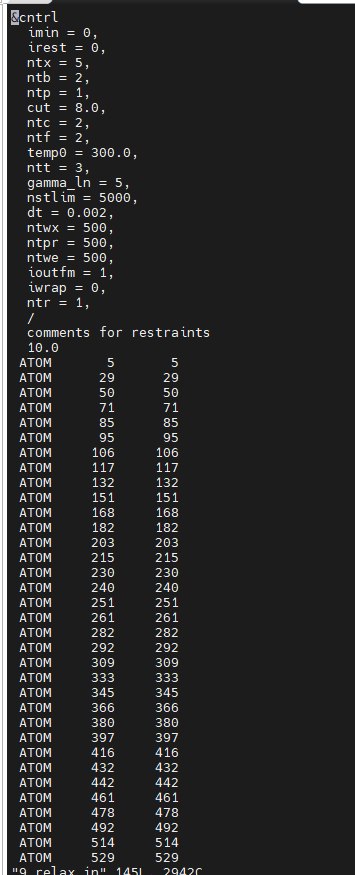 |
&cntrl imin = 0, irest = 0, ntx = 5, ntb = 2, ntp = 1, cut = 8.0, ntc = 2, ntf = 2, temp0 = 300.0, ntt = 3, gamma_ln = 5, nstlim = 5000, dt = 0.002, ntwx = 500, ntpr = 500, ntwe = 500, ioutfm = 1, iwrap = 0, ntr = 1, / comments for restraints 10.0 #该部分为所有“CA” 原子的atom number,此处仅为两个原子的实例 ATOM 5 5 ATOM 120 120 END END |
(每一标亮部分都有8个letter,通过命令:grep CA OCT_complex_human_E-selectin_sLex_WAT.pdb > restraint.txt获取,再通过vim编辑器编辑成要求格式)
10.produ.in
&cntrl imin = 0, irest = 0, ntx = 5, ntb = 2, ntp = 1, cut = 8.0, ntc = 2, ntf = 2, temp0 = 300.0, ntt = 3, gamma_ln = 5, nstlim = 5000000, dt = 0.002, ntwx = 500, ntpr = 500, ntwe = 500, ntwr = 500, ntwprt = 2060, ioutfm = 1, iwrap = 0, / END
参数ntwprt为实际的原子个数,包含Na+/Cl-,不包含水,该参数需根据自己文件修改
4.准备可执行文件
在 md_simulations目录下新建文件run.sh,将以下内容粘贴至run.sh文件中。其中,标绿的文件为输入文件,标蓝的文件为输出文件。
run.sh
#step 1 mpirun -np 28 pmemd.MPI -O -i 1.min.in -o 1.min.o -p OCT_complex_human_E-selectin_sLex_WAT.prmtop -c OCT_complex_human_E-selectin_sLex_WAT.rst7 -r 1.min_complex_human_E-selectin_sLex_WAT.rst7 -ref OCT_complex_human_E-selectin_sLex_WAT.rst7 #step 2 mpirun -np 28 pmemd.MPI -O -i 2.relax.in -o 2.relax.o -p OCT_complex_human_E-selectin_sLex_WAT.prmtop -c 1.min_complex_human_E-selectin_sLex_WAT.rst7 -r 2.relax_complex_human_E-selectin_sLex_WAT.rst7 -ref 1.min_complex_human_E-selectin_sLex_WAT.rst7 #step 3 mpirun -np 28 pmemd.MPI -O -i 3.min.in -o 3.min.o -p OCT_complex_human_E-selectin_sLex_WAT.prmtop -c 2.relax_complex_human_E-selectin_sLex_WAT.rst7 -r 3.min_complex_human_E-selectin_sLex_WAT.rst7 -ref 1.min_complex_human_E-selectin_sLex_WAT.rst7 #step 4 mpirun -np 28 pmemd.MPI -O -i 4.min.in -o 4.min.o -p OCT_complex_human_E-selectin_sLex_WAT.prmtop -c 3.min_complex_human_E-selectin_sLex_WAT.rst7 -r 4.min_complex_human_E-selectin_sLex_WAT.rst7 -ref 1.min_complex_human_E-selectin_sLex_WAT.rst7 #step 5 mpirun -np 28 pmemd.MPI -O -i 5.min.in -o 5.min.o -p OCT_complex_human_E-selectin_sLex_WAT.prmtop -c 4.min_complex_human_E-selectin_sLex_WAT.rst7 -r 5.min_complex_human_E-selectin_sLex_WAT.rst7 #step 6 mpirun -np 28 pmemd.MPI -O -i 6.relax.in -o 6.relax.o -p OCT_complex_human_E-selectin_sLex_WAT.prmtop -c 5.min_complex_human_E-selectin_sLex_WAT.rst7 -r 6.relax_complex_human_E-selectin_sLex_WAT.rst7 -ref 5.min_complex_human_E-selectin_sLex_WAT.rst7 #step 7 mpirun -np 28 pmemd.MPI -O -i 7.relax.in -o 7.relax.o -p OCT_complex_human_E-selectin_sLex_WAT.prmtop -c 6.relax_complex_human_E-selectin_sLex_WAT.rst7 -r 7.relax_complex_human_E-selectin_sLex_WAT.rst7 -ref 5.min_complex_human_E-selectin_sLex_WAT.rst7 #step 8 mpirun -np 28 pmemd.MPI -O -i 8.relax.in -o 8.relax.o -p OCT_complex_human_E-selectin_sLex_WAT.prmtop -c 7.relax_complex_human_E-selectin_sLex_WAT.rst7 -r 8.relax_complex_human_E-selectin_sLex_WAT.rst7 -ref 5.min_complex_human_E-selectin_sLex_WAT.rst7 #step 9 mpirun -np 28 pmemd.MPI -O -i 9.relax.in -o 9.relax.o -p OCT_complex_human_E-selectin_sLex_WAT.prmtop -c 8.relax_complex_human_E-selectin_sLex_WAT.rst7 -r 9.relax_complex_human_E-selectin_sLex_WAT.rst7 -ref 5.min_complex_human_E-selectin_sLex_WAT.rst7 mpirun -np 28 pmemd.MPI -O -i 10.produ.in -o 10.produ.o -p OCT_complex_human_E-selectin_sLex_WAT.prmtop -c 9.relax_complex_human_E-selectin_sLex_WAT.rst7 -r 10.produ_complex_human_E-selectin_sLex_WAT.rst7 -x produ_complex_human_E-selectin_sLex_WAT.nc